در این مقاله می آموزید:
- نحوه ی خواندن داده های متا از یک فایل DICOM
- نحوه ی خواندن داده های عکس، از یک فایل DICOM
- نحوه ی نوشتن داده های عکس یا داده های متا در یک فایل DICOM
- نحوه ی استفاده از فرمت Mayo Analyze 7.5
- نحوه ی استفاده از فرمت Interfile
خواندن داده های متا از یک فایل DICOM
فایل های DICOM حاوی یک سری داده های متا هستند. این داده های متا، درواقع یک سری اطلاعات در مورد عکس مورد نظر هستند. بعنوان مثال، داده های متا حاوی اطلاعاتی از قبیل سایز، ابعاد، عمق بیت، تنظیمات عکس برداری و غیره هستند.
برای خواندن داده های متا از یک فایل DICOM، می توانید از تابع dicominfo استفاده کنید. تابع dicominfo اطلاعات مورد نظر را در قالب یک ساختار(در متلب) برمی گرداند. هر فیلد در این ساختار، مشخص کننده ی یک بخش از داده های متای این فایل DICOM است. شما می توانید از این ساختار داده های متا که توسط تابع dicominfo برگردانده شده است در یک فایل DICOM که توسط تابع dicomread خوانده می شود، استفاده کنید.
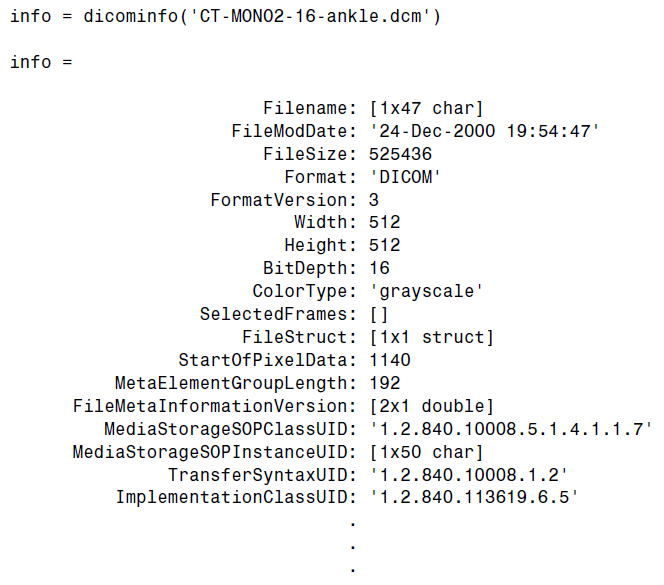
در مثال زیر، داده های متا از یک فایل نمونه ی DICOM که در جعبه ابزار متلب قرار دارد، خوانده می شوند:
خواندن داده های عکس از یک فایل DICOM
برای خواندن داده های عکس از یک فایل DICOM، می توانید از تابع dicomread استفاده کنید. تابع dicomread به خصوصیات فرمت DICOM مربوط می شود اما قادر است تا بعضی از فایل های متداول غیر مرتبط را نیز بخواند. هنگامی که از تابع dicomread استفاده می کنید، می توانید مثل مثال زیر، نام فایل مورد نظر را بصورت یک آرگومان مشخص کنید. مثال زیر، یک فایل نمونه ی DICOM که در جعبه ابزار متلب قرار دارد را می خواند.
مثال (خواندن داده های پزشکی در پردازش تصویر)
info = dicominfo('CT-MONO2-16-ankle.dcm');
I = dicomread(info);
مشاهده ی عکس های داخل یک فایل DICOM
برای مشاهده ی داده های یک عکس که از یک فایل DICOM خوانده شده اند، می توانید از توابع imshow یا imtool استفاده کنید. اما چون که داده های این فایل DICOM به صورت 16 بیتی علامت دار هستند، شما باید از سینتکس مقیاس دهی خودکار در هر یک از توابع ذکر شده استفاده کنید تا بتوانید این عکس ها را مشاهده کنید:
مثال (خواندن داده های پزشکی در پردازش تصویر)
imshow(I,'DisplayRange',[])
نوشتن داده های عکس یا داده های متا در یک فایل DICOM
برای نوشتن داده های عکس یا داده های متا در یک فایل با فرمت DICOM می توانید از تابع dicomwrite استفاده کنید. در مثال زیر، عکس I را در داخل فایلی با فرمت DICOM به نام ankle.dcm می نویسیم:
مثال (خواندن داده های پزشکی در پردازش تصویر)
dicomwrite(I,'ankle.dcm')
نوشتن داده های متا همراه با داده های عکس
هنگامی که داده های یک عکس را در داخل یک فایل DICOM می نویسیم، دستور dicomwrite به طور اتوماتیک مجموعه ای از فیلدهای ضروری داده های متا را که از نوع IOD هستند به فایل مورد نظر اضافه می کند. دستور dicomwrite از موارد زیر به طور کامل پشتیبانی می کند:
- ضبط ثانویه(Secondary capture)
- تشدید مغناطیسی(Magnetic resonance)
- توموگرافی کامپیوتری(Computed tomography)
دستور dicomwrite قادر است تا انواع دیگر داده های DICOM را نیز تولید کند( مانند پرتو درمانی اشعه ایکس یا پرتو درمانی هسته ای). اما این دستور صحت این داده ها را تایید نمی کند.
شما همچنین می توانید داده های متایی را که از قبل با استفاده ازدستور dicominfo به دست آورده اید را به تابع dicomwrite بدهید. در مثال زیر، تابع dicomwrite داده های متای قرار گرفته در متغیر info را به یک فایل DICOM جدید اضافه می کند.
مثال (خواندن داده های پزشکی در پردازش تصویر)
info = dicominfo('CT-MONO2-16-ankle.dcm');
I = dicomread(info);
dicomwrite(I,'ankle.dcm',info) توجه کنید که داده های متای نوشته شده در این فایل با داده های متای متغیر info یکسان نستند. هنگامی که می خواهیم داده های متا را در یک فایل بنویسیم، بعضی از فیلدها باید توسط دستور dicomwrite آپدیت شوند. برای فهمیدن این موضوع به مقدار فیلد ID در داده های متای اصلی و در فایل جدید ایجاد شده نگاهی بیاندازید:
مثال (خواندن داده های پزشکی در پردازش تصویر)
info.SOPInstanceUID
ans =
1.2.840.113619.2.1.2411.1031152382.365.1.736169244
اکنون داده های متای آن فایل DICOM که به تازگی ایجاد شده است را با استفاده از دستور dicominfo می خوانیم. و سپس مقدار فیلد SOPInstanceUID را مورد بررسی قرار می دهیم. همان طور که مشاهده می کنید، بعضی از مقادیر آن، متفاوت هستند:
مثال (خواندن داده های پزشکی در پردازش تصویر)
info2 = dicominfo('ankle.dcm');
info2.SOPInstanceUID
ans =
1.2.841.113411.2.1.2411.10311244477.365.1.63874544
حذف اطلاعات محرمانه از یک فایل DICOM
هنگام استفاده از یک فایل DICOM در زمینه ی آموزش یا مطالعات تحقیقاتی، ممکن است بخواهید اطلاعات محرمانه ی خود را از آن فایل حذف کنید. برای انجام این کار می توانید ار تابع dicomanon استفاده کنید.
تابع dicomanon با استفاده از مقادیر جدید به دست آمده از مطالعات، یک دنباله ایجاد می کند و سپس برخی از داده های متا را تغییر می دهد و سپس فایل مورد نظر را تولید می کند(می نویسد).
مثال:ایجاد یک دنباله ی جدید DICOM
هنگامی که یک عکس اصلاح شده را در یک فایل DICOM می نویسید، ممکن است بخواهید که آن عکس های اصلاح شده بصورت یک دنباله باشند. در فرمت استاندارد DICOM، عکس ها می توانند بصورت یک دنباله، مرتب شوند. هنگامی که شما یک عکس را به همراه داده های متا در یک فایل DICOM می نویسید، دستور dicomwrite این عکس را به طور پیش فرض در دنباله ی قبلی قرار می دهد. برای ایجاد یک دنباله ی جدید، باید یک شناسه ی یکتای جدید را به فیلد متا دیتای SeriesInstanceUID انتساب دهید. در مثال زیر روش این کار نشان داده شده است:
1. ابتدا یک عکس از نوع DICOM را در پنجره ی Workspace متلب، بخوانید.
مثال (خواندن داده های پزشکی در پردازش تصویر)
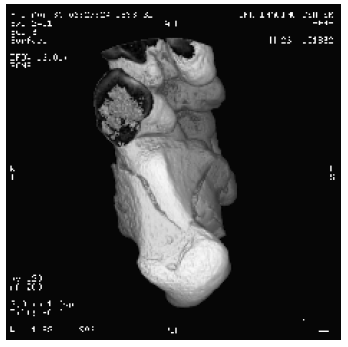
I = dicomread('CT-MONO2-16-ankle.dcm'); حالا برای اینکه بتوانید این عکس را مشاهده، می توانید از تابع imshow یا imtool استفاده کنید. به دلیل اینکه داده های عکس DICOM به صورت 16 بیتی علامت دار هستند، باید از سینتکس مقیاس دهی خودکار استفاده کنید:
مثال (خواندن داده های پزشکی در پردازش تصویر)
imtool(I,'DisplayRange',[])
نتیجه به صورت زیر خواهد بود:

2. داده های متا را از فایل DICOM قبلی بخوانید:
مثال (خواندن داده های پزشکی در پردازش تصویر)
info = dicominfo('CT-MONO2-16-ankle.dcm'); برای مشخص کردن اینکه این عکس، به کدام دنباله تعلق دارد، مقدار فیلد SeriesInstanceUID را مشاهده کنید:
مثال (خواندن داده های پزشکی در پردازش تصویر)
info.SeriesInstanceUID
ans =
1.2.840.113619.2.1.2411.1031152382.365.736169244
3: شما باید تنها هنگامی که می خواهید به طریقی عکس مورد نظر خود را اصلاح کنید، یک دنباله ی جدید DICOM را ایجاد کنید. در مثال زیر تمام متن ها را از عکس خود حذف می کنیم.
در این مثال، ابتدا ماکسیمم و مینیمم تمام پیکسل های درون عکس خود را پیدا می کنیم. پیکسل هایی که به رنگ سفید هستند، مقادیر ماکسیمم را مشخص می کنند.
مثال (خواندن داده های پزشکی در پردازش تصویر)
max(I(:))
ans =
4080
min(I(:))
ans =
32
برای حذف این کاراکترهای متنی، تمام پیکسل های ماکسیمم را برابر با مقدار پیکسل های مینیمم قرار می دهیم:
مثال (خواندن داده های پزشکی در پردازش تصویر)
Imodified = I;
Imodified(Imodified == 4080) = 32;
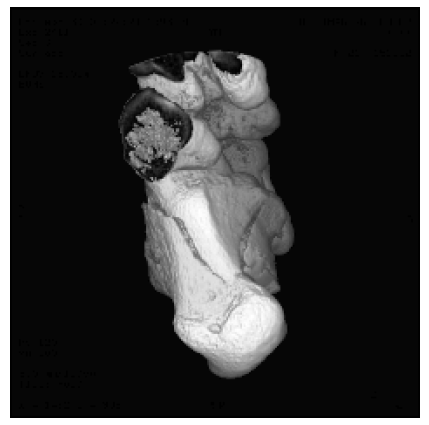
حالا با دستور زیر عکس خود را مشاهده می کنیم.
مثال (خواندن داده های پزشکی در پردازش تصویر)
imshow(Imodified,[])
عکس ما به صورت زیر در خواهد آمد:
4.حالا یک شناسه ی یکتای DICOM که به آن UID می گوییم را با استفاده از تابع dicomuid تولید کنید. شما برای نوشتن این عکس اصلاح شده در یک دنباله ی جدید، به یک UID جدید احتیاج دارید.
مثال (خواندن داده های پزشکی در پردازش تصویر)
uid = dicomuid
uid =
1.3.6.1.4.1.9590.100.1.1.56461980611264497732341403390561061497
تابع dicomuid یک UID یکتای جدید را ایجاد می کند.
5.حالا مقدار فیلد SeriesInstanceUID در داخل داده های متای عکس DICOM اصلی(عکس اورجینال) را برابر با این مقدار تولید شده قرار دهید.
مثال (خواندن داده های پزشکی در پردازش تصویر)
info.SeriesInstanceUID = uid;
6. حالا آن عکس اصلاح شده را در یک فایل DICOM جدید بنویسید. و داده های متای اصلاح شده، یعنی متغیر info را بعنوان یک آرگومان به آن بدهید. به دلیل اینکه شما مقدار SeriesInstanceUID را تنظیم کرده اید، عکسی که اکنون ایجاد می کنید، به یک دنباله ی جدید تعلق دارد.
مثال (خواندن داده های پزشکی در پردازش تصویر)
dicomwrite(Imodified,'ankle_newseries.dcm',info);
برای اینکه از صحت عملیات انجام شده اطمینان حاصل کنید، داده های متای فیلد SeriesInstanceUID را در فایل بالا مشاهده کنید.
استفاده از فرمت Mayo Analyze 7.5
فرمت Analize 7.5 توسط فردی به نام Mayo Clinic برای ذخیره ی داده های MRI توسعه داده شده است. مجموعه داده های Analize 7.5 شامل دو نوع فایل هستند:
- فایل عنوان (مثلا بصورت filename.hdr)
این فایل اطلاعاتی را در مورد ابعاد و نحوه ی شناسایی و تاریخ پردازش تصویر، ارائه می دهد. شما می توانید از تابع analyze75info برای خواندن اطلاعات از فایل عنوان، استفاده کنید.
2. فایل تصویر(مثلا filename.img)
این فایل اطلاعات عکسی که نوع داده های آن و ترتیب قرار گرفتن آنها توسط فایل عنوان تعیین شده است را مشخص می کند. شما می توانید از تابع analyze75read برای خواندن داده های این عکس ها استفاده کنید.
مثال زیر، از تابع analyze75info برای خواندن داده های متا، از یک فایل عنوان Analyze 7.5 استفاده می کند، سپس اطلاعات برگردانده شده از تابع analyze75info را به تابع analyze75read می دهد تا عکس مورد نظر را خوانده شود.
مثال (خواندن داده های پزشکی در پردازش تصویر)
info = analyze75info('CT_HAND.hdr');
X = analyze75read(info); استفاده از فرمت Interfile
از فرمت Interfile برای تبادل داده های عکس های پزشکی هسته ای استفاده می شود. یک مجموعه از داده ها با فرمت Interfile حاوی دو فایل است:
- فایل عنوان( مثل filename.hdr): این فایل اطلاعاتی را در مورد ابعاد و نحوه ی شناسایی و تاریخ پردازش تصویر، ارائه می دهد. شما می توانید از تابع interfileinfo برای خواندن داده های عنوان، استفاده کنید.
- فایل عکس(مثل filename.img): این فایل داده های عکسی که نوع داده های آن و ترتیب قرار گرفتن آنها توسط فایل عنوان تعیین شده است را مشخص می کند. شما می توانید از تابع interfileread برای خواندن داده های این عکس در متلب استفاده کنید.
مثال زیر، تابع interfileinfo را فراخوانی می کند تا داده های متا را از فایل عنوان بخواند. سپس در این مثال، از عکس مربوطه، داده های عکس مورد نظر را می خوانیم.
مثال (خواندن داده های پزشکی در پردازش تصویر)
info = interfileinfo('dyna');
X = interfileread('dyna');







خداقوت
حرف نداشت
واقعا احسنت
با این که هیچ وقت کامنت نمیدادم اونقد تحسین داشت که نمیشد چیزی نگم
تشکر